| エピジェネティクスとは? | |
| エピジェネティクスと疾患 | |
|
|
| Hpa II認識部位のゲノムでの分布と数 | |
| MIAMI法の手技 | |
| MIAMI法の解析例 | |
| MIAMI法のプロトコール |
| MIAMI法(マイクロアレイによるゲノムワイドなメチル化解析法)とは? |
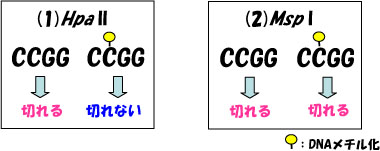
| 癌をはじめ様々な疾患にエピジェネティクス、DNAメチル化が関与することからゲノム全体にわたってDNAメチル化を調べることの重要性が認識されるようになってきています。このような方法として我々は1991年にRestriction Landmark Genomic Scanning (RLGS)法(文献1、文献2)を開発しましたが、すべての遺伝子を調べられないことや手技が煩雑であるなどといった欠点がありました。そこでこのような欠点を克服したMicroarray-based Integrated Analysis of Methylation by Isoschizomers (MIAMI)法(文献3)を開発しました。この方法ではDNAメチル化の検出に制限酵素を用います。DNAのメチル化はシトシン塩基の5の位置で起こることが知られていますが、そのほとんどがCGという配列のシトシンに集中していることがわかっています。Hpa IIという制限酵素はCGを含むCCGGという配列を認識して切断しますが、内側のCがメチル化されているとDNAを切断できません(メチル化感受性酵素) 。一方Hpa IIと同じCCGG配列を認識する制限酵素にMsp Iがあります。この酵素はHpa IIと異なり内側のCがメチル化されていてもされていなくてもDNAを切断することができます(メチル化非感受性酵素)。MIAMI法では、比較するサンプル間で(1)Hpa IIによる切れ具合の差と同時に(2)Msp Iによる切れ具合の差を比較することによりサンプル間のメチル化の差を検出します。(1)Hpa IIの切れ具合の差はサンプル間のメチル化の差と関係していますが、(2)Msp Iによる切れ具合の差も測定することにより信頼性を検定することができます。もしサンプル間に多型や質の差があればメチル化の差がなくても(1)Hpa IIの切れ具合の差が観察されるが、この場合は(2)Msp Iによる切れ具合の差も観察されるのでfalse positiveと判定することができる。 |
| 問い合わせ先 群馬大学生体調節研究所附属 生体情報ゲノムリソースセンター 畑田出穂 hatada@gunma-u.ac.jp |
|
Link
|
| 群馬大学 生体情報ゲノムリソースセンター |
| ゲノム科学リソース分野(畑田研) |
| 群馬大学 |
| 群馬大学 生体調節研究所 |
| 群馬大学 医学部 |
| 群馬大学 教育学部 |
| 群馬大学 社会情報学部 |
| 群馬大学 工学部 |
| 全国遺伝子実験施設 |
もっと詳しく知りたい人はこちら→

