研究内容
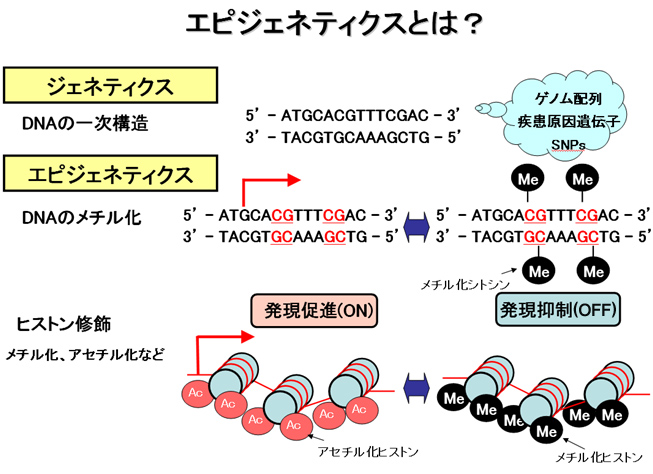
エピジェネティクスとは?
私たちの研究室では“エピジェネティクス”という現象の研究をおこなっております。エピジェネティクスとは遺伝情報が同じでも生物の表現型が同じにならない原因です。例えば一卵性双生児やクローン動物がかならずしも同じではなかったり、我々の体が若いときと年をとったときで異なるのもエピジェネティクスが原因だといわれています。エピジェネティクスの実体はDNAのメチル化などのゲノムの印付けであり“エピゲノム”といいますが、これが遺伝子のスイッチとなってオン、オフすることにより同じ遺伝子を持っていても異なる結果をもたらしています。エピジェネティクスはポストゲノム時代における重要な研究テーマであり、この研究によりこれまでのゲノム研究で明らかにはできなかった生命現象や疾患の原因が明らかになると言われています。私たちの研究室ではエピジェネティクスの視点で様々な生命現象、癌、生活習慣病、再生医療といった疾患の研究に取り組んでおります。
エピゲノムと疾患
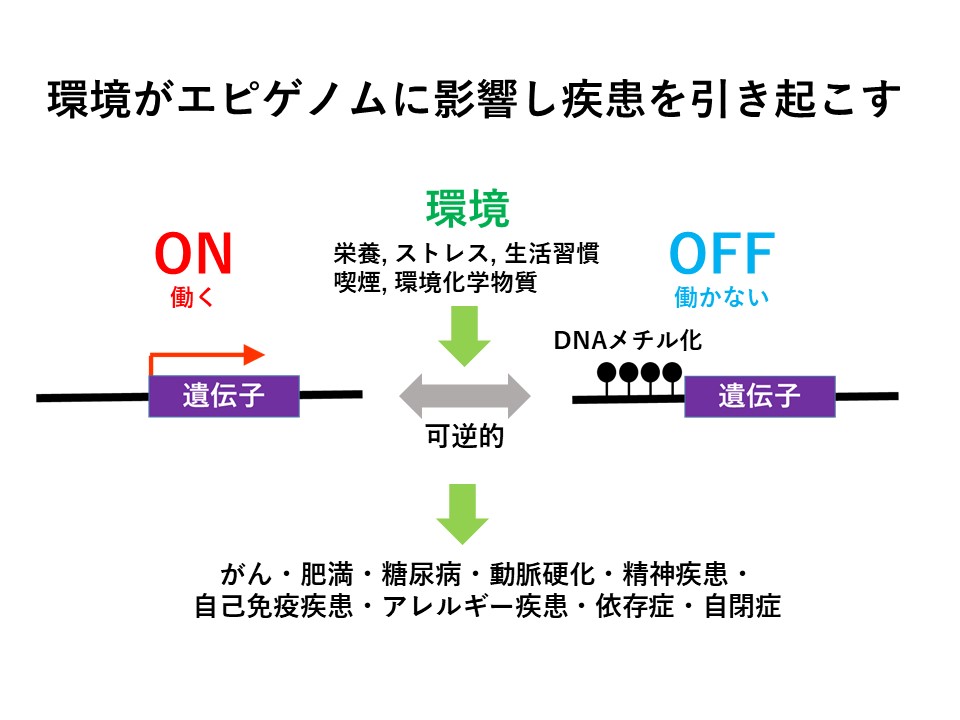
エピゲノムは栄養状態、ストレス、生活習慣、喫煙、環境化学物質など様々な環境の影響を受けて変化します。その結果、がん、肥満、糖尿病、動脈硬化、精神疾患、自己免疫疾患、アレルギー疾患、依存症、自閉症など様々な疾患を引き起こします。例えばがんでは発がんを抑える働きを持つがん抑制遺伝子メチル化されオフになっていることが知られております。
エピゲノムを操作は治療につながる – エピゲノム編集法の開発
エピゲノムの異常により疾患が発症するのであれば、患者のエピゲノムを操作できれば治療につながると考えられます。
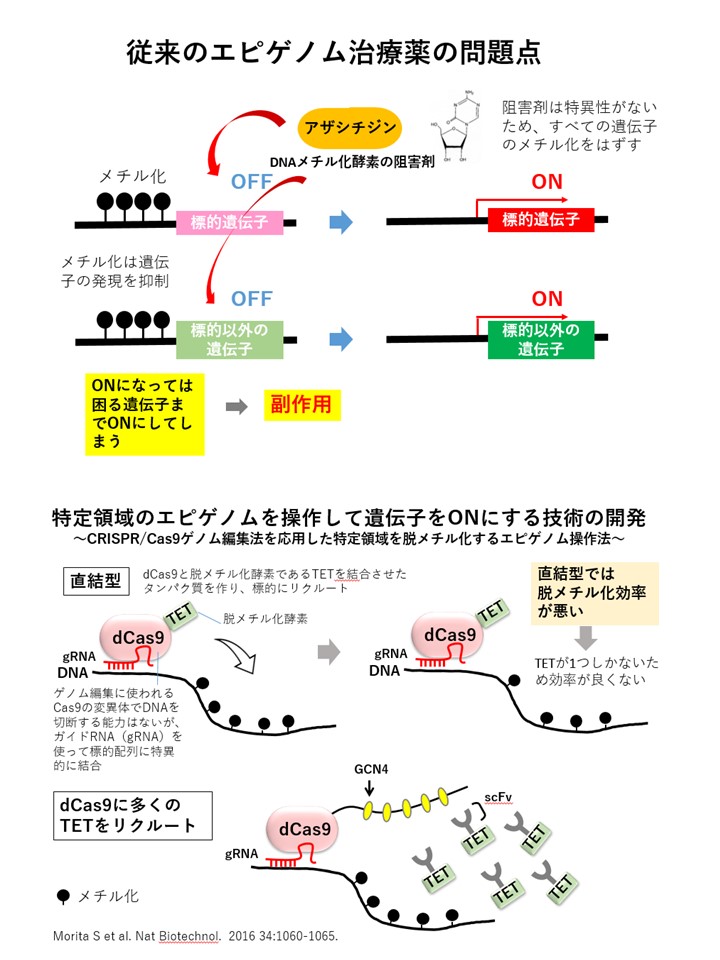
従来、エピゲノムの操作にはアザシチジンなどのDNAメチル化酵素の阻害剤が使われていました。しかし、この阻害剤は特異性がなく、標的とする遺伝子だけでなく、それ以外の遺伝子のメチル化も解除してしまい、結果としてすべての遺伝子のスイッチがオンになるという問題がありました。そのため、副作用が生じるリスクがありました。
この問題を解決するために、特定の遺伝子のエピゲノムだけを操作できる技術の開発が求められていました。
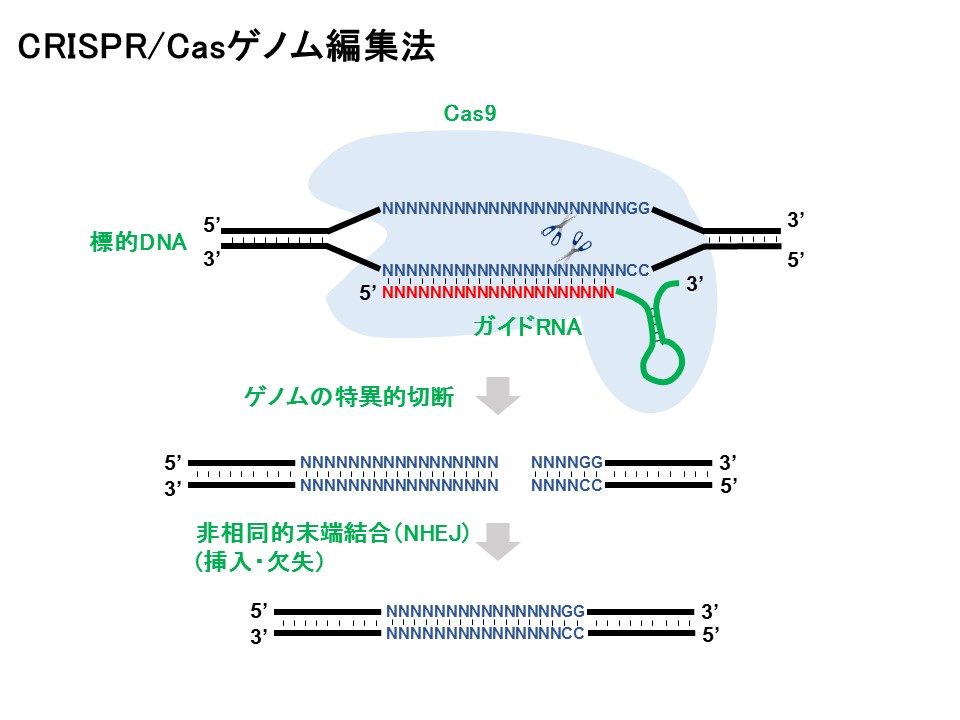
そこで、私たちはゲノム編集技術CRISPR/Cas9(クリスパー キャスナイン)を応用し、特定の領域(狙った遺伝子)のメチル化を解除して遺伝子をオンにするエピゲノム編集技術を開発しました(Morita et al. 2016)。
私たちが使用しているのは、Cas9という酵素の変異型であるdCas9です。通常のCas9は標的のDNAを切断しますが、dCas9を利用するとDNAメチル化を解除する酵素を呼び寄せ、狙った遺伝子のスイッチをオンにすることができます。非常に便利な技術です。
dCas9はガイドRNAを用いることで、標的のDNAに特異的に結合できます。そこで、dCas9に脱メチル化酵素であるTETを結合させ、それを細胞に導入しました。
しかし、TETが1つしかないためか、脱メチル化の効率があまり良くありませんでした。そこで、多くのTETを標的にリクルートする技術を開発しました。そのために、GCN4という短いペプチドと、それを特異的に認識する一本鎖抗体(scFv)を利用しました。scFvは、抗体を小型化したミニ抗体のようなものです。
私たちは、dCas9の後方に複数のGCN4を付加したものと、TETとscFvを融合させたものを使用し、標的に多くのTETをリクルートすることで、効率的に脱メチル化を行い、遺伝子をオンにすることに成功しました。
この技術を用いて疾患でオフになってしまっている遺伝子をオンにすることが可能になり、疾患の治療への応用が期待されます。
- Morita S, Noguchi H, Horii T, Nakabayashi K, Kimura M, Okamura K, Sakai A, Nakashima H, Hata K, Nakashima K, Hatada I. Targeted DNA demethylation in vivo using dCas9–peptide repeat and scFv–TET1 catalytic domain fusions. Nature Biotechnology 2016 Oct;34(10):1060-1065. doi: 10.1038/nbt.3658.
エピゲノム疾患モデルマウス
疾患治療研究において疾患モデル動物は欠かせません。治療法をヒトでいきなり試すのは危険なので、一般にヒトの疾患と同じ症状を示す動物(疾患モデル動物)を用いて治療研究がまず行われます。これまで遺伝子改変技術を用いて遺伝子を改変することにより遺伝病の疾患モデル動物をつくることはできましたが、エピゲノム疾患のモデル動物は存在しませんでした。そこで我々の開発したエピゲノム編集法をマウスに応用してエピゲノム疾患モデルマウスを作製することに成功しました(Horii et al. 2020)。
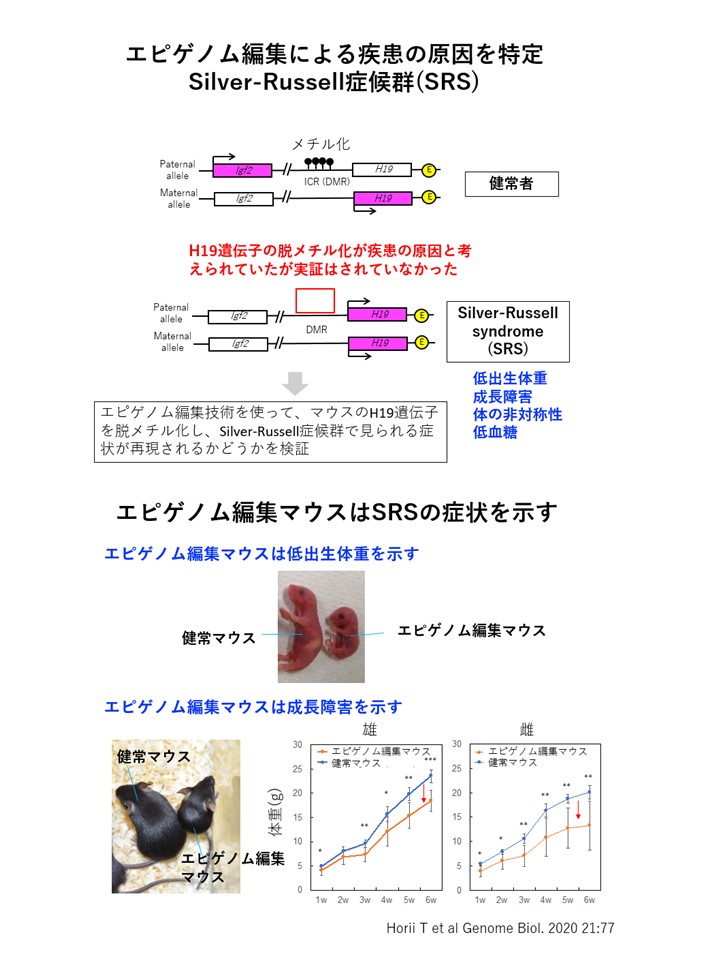
シルバー・ラッセル症候群(SRS)は、低出生体重、成長障害、体の非対称性、低血糖などの症状を示す疾患です。その原因は、H19という遺伝子が脱メチル化することだと考えられていましたが、これまで実証されていませんでした。
そこで、エピゲノム編集技術を使って、マウスのH19遺伝子を脱メチル化して、シルバー・ラッセル症候群で見られる症状が再現されるかどうかを検証しました。その結果、エピゲノム編集を行ったマウスではSRSと同様に低出生体重と成長障害、低血糖などをはじめとする症状が再現されました。 このようにエピゲノム編集マウスでシルバー・ラッセル症候群の症状が再現することから、H19遺伝子の脱メチル化がこの疾患の原因であることが証明されました。つまり、エピゲノム編集は疾患の原因遺伝子を特定するために利用できることがわかりました。またこのマウスはシルバー・ラッセル症候群のモデルマウスとして治療研究に用いることが可能です。
- Horii T, Morita S, Hino S, Kimura M, Hino Y, Kogo H, Nakao M & Hatada I. Successful generation of epigenetic disease model mice by targeted demethylation of the epigenome.Genome Biology 2020 Apr 1;21(1):77. doi: 10.1186/s13059-020-01991-8. PubMed PMID:32198422; PubMed Central PMCID: PMC7083849.
- Horii T, Morita S, Kimura M, Hatada I. Efficient generation of epigenetic disease model mice by epigenome editing using the piggyBac transposon system. Epigenetics Chromatin 2022 Dec 16;15(1):40. doi: 10.1186/s13072-022-00474-3.
CRISPR/Casゲノム編集法を用いた疾患モデルマウス、疾患モデルiPS細胞研究
CRISPR/Casは効率がよく簡便なゲノム編集システムです。このシステムではガイドRNAというゲノム中の標的と相補的な短いRNAとCas9というDNA切断酵素の複合体が標的を切断することにより高効率にノックアウト細胞を作製することができます。このシステムを用いてエピジェネティクス関連遺伝子が関与する疾患モデルを作製し、研究をおこなっています。方法には2通りあり、その1つはCRISPR/Casゲノム編集で疾患モデル動物を作製する方法です(Horii et al. 2014)。またヒト細胞における表現型を調べたいときはiPS細胞の遺伝子を改変することにより疾患モデルiPS細胞を作製して研究に用いています(Horii et al. 2013)。このようにして作製した疾患モデルiPS細胞は患者から作製したものと異なり、作製の元になった正常人由来のiPS細胞をコントロールとして研究にもちいれば遺伝的背景の違いによる表現型の違いがないので非常に有用です。
- Horii T, Arai Y, Yamazai M, Morita S, Kimura M, Itoh M, Abe Y, Hatada I. Validation of microinjection methods for generating knockout mice by CRISPR/Cas-mediated genome engineering. Scientific Reports, 4:4513, 2014 詳しくは、日経バイオテク
- Horii T, Tamura D, Morita S, Kimura M, Hatada I. Generation of an ICF Syndrome Model by Efficient Genome Editing of Human Induced Pluripotent Stem Cells Using the CRISPR System. Int J Mol Sci,14: 19774-19781, 2013 詳しくは 紹介記事